Ácidos Nucleicos recombinantes en la naturaleza
La β-glucosidasa
recombinante es una clase de enzima que se distribuye ampliamente en el
mundo viviente, con ejemplos en plantas, hongos, animales y bacterias. Ofrecen
capacidad tanto de hidrólisis como de síntesis para una amplia gama de procesos
biotecnológicos. Sin embargo, la disponibilidad de β-glucosidasas nativas es
actualmente un cuello de botella en la aplicación industrial generalizada de
esta enzima. (1)
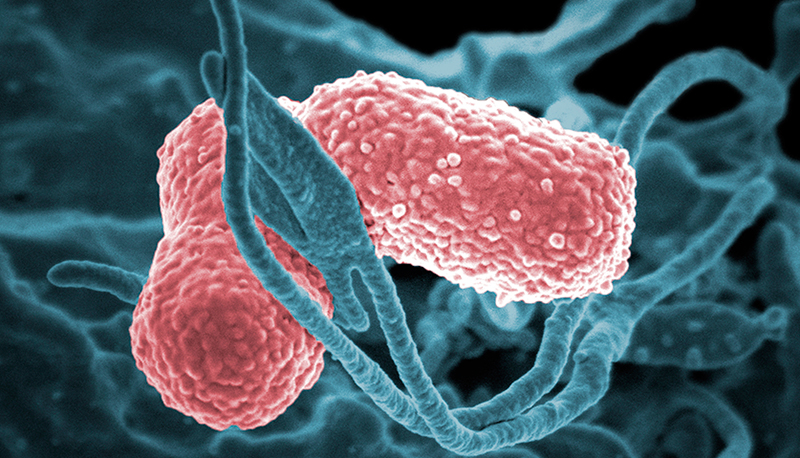
ADN recombinante artificial de la Enfermedad infecciosa por Enterobacter cloacae
T: Caracterización genética y bioquímica de FRI-1, una β-lactamasa de clase A hidrolizante de carbapenémicos de Enterobacter cloacae
O: Caracterizar a nivel genético y bioquímico los mecanismos moleculares de resistencia a carbapenémicos de un aislado de Enterobacter cloacae
G: gen FRI-1 bla
ER: Sau3AI, BamHI
EL: Plásmido pBK-CMV
V: pBK-CMV resistente a kanamicina
CR: E. coliTOP10
MTG: Electroporación
MIC: Cultivó (pFRI) durante la noche a 37ºC en 2 litros de caldo TS que contenía ticarcilina (100 µg / ml) y kanamicina (50 µg / ml), y amplificación por PCR.





:quality(85)//s3.amazonaws.com/arc-wordpress-client-uploads/infobae-wp/wp-content/uploads/2017/01/17151206/iStock-499775068.jpg)



